BIOSAFETY ASSESSMENT AND MANAGEMENT OF GENOME EDITING ASSISTED BREEDING OF FARMED FISH
-
摘要:
针对特定遗传位点实现定点突变的基因编辑技术的出现, 大大加速农业生物育种的进程。文章在简述基因编辑技术在作物、牲畜性状改良带来的快速、颠覆性改良研究与应用的基础上, 着重关注国内外学者对养殖鱼类的基因编辑技术辅助育种方面的研究。在回顾了我国学者在养殖鱼类生产与品质性状改良的亮点成果之后, 文章重点讨论了国内外学者对养殖鱼类基因编辑育种的生物安全, 包括食品安全和生态安全的论点; 并对基因编辑鱼类导致食品安全和生态安全风险的评价、防控策略等研究进展进行论述。最后, 对“可控不育”技术研究进展, 及其在基因编辑养殖鱼类生态安全风险防控、育种知识产权保障、编辑鱼类的产业化应用的促进作用和未来发展进行了展望。
Abstract:The emergence of gene editing to make precise and targeted changes to the genetic code of plants and animals has attracted immense attention for its ability to create new varieties with specific traits more efficiently and accurately than traditional breeding methods. After a brief review of the amazing effects of genome-edited breeding in crops and livestock, this review has focused mainly on studies of the application of gene editing in farmed fish. It has been found that the global advantage of trait improvement in farmed fish in China has been achieved. In the present review, more attention has been paid to the biosafety assessment and management, including food safety and ecological safety, caused by gene edited farmed fish. At last, based on the progress of various techniques of “controllable sterility”, its significant roles on ecological risk prevention, intellectual property protection and marketing promotion of gene edited farmed fish have been introduced and prospected.
-
Keywords:
- Gene editing /
- Farmed fish breeding /
- Biosafety /
- Controllable sterility
-
进入21世纪以来, 水产食品承担了更大比例的人类营养需求, 根据世界粮农组织《2024年全球渔业与水产养殖状况》报告, 全球养殖水产品产量在2022年首次超过了捕捞渔业产量, 显示未来养殖模式将是水产食品日益重要的来源[1]。2022年, 世界鱼类养殖产量占水产动物养殖量的75%, 而淡水鱼类养殖约占世界鱼类养殖量的83%[1]。因此, 鱼类养殖, 尤其是淡水鱼类养殖对于保障全球动物蛋白食品供应具有重要意义。面对全球人口增长、气候变化等因素, 以及水产业发展与水体环境的不可分割的运行特征, 推动水产食品系统的可持续发展时需要考虑维护好水体生态系统健康, 如减少富含营养物质的养殖尾水排放、保护水体生物多样性等多种迫切关切。为此, 利用新型高效精准育种技术, 改良养殖鱼类经济性状, 如饲料利用率提升、耐高温低氧、具高不饱和脂肪酸与无肌间刺等等, 从而提供减少对自然资源的依赖、满足消费者对品质的要求、改善养殖生物对环境变化适应等新养殖对象, 不仅可以促进社会平等、公平和稳定发展, 还使水产养殖产业真正成为人类粮食和营养安全, 以及维护地球生态安全的重要支撑。
本文将针对基因编辑(Gene Editing)辅助的鱼类育种技术在养殖鱼类关键生产、品质性状的颠覆性改良效果, 以及潜在生态安全风险进行文献综述, 并对基因编辑辅助应用于鱼类育种的生物安全评价与管控策略进行探讨。本文还将围绕鱼类育性可控技术的研究意义和进展进行介绍, 并对未来基因编辑辅助鱼类育种的应用前景进行展望。
1. 基因编辑技术及其在生物育种中的应用
1.1 基因编辑技术
基因编辑是指通过特定技术, 实现对生物体基因组进行修改和调整的过程。这项技术近年来取得了显著进展, 如CRISPR-Cas9技术可以高效、精准对特定基因位点核苷酸碱基插入、缺失或替换, 使特定位点的DNA序列发生改变, 构建可遗传的表型改变的生物品系。
基因编辑技术研究始于20世纪80年代末, 研究发现对诱导的基因组DNA特定位点的双链断裂(Double-stranded break, DSB)损伤伴生的修复, 可导致在该位点发生染色体水平上细微改变。当DSB被诱导后, 细胞内将启动两种主要的修复机制: 同源重组(Homology directed repair, HDR)和非同源末端连接(Nonhomologous end joining, NHEJ)。其中, 同源重组依赖于DNA模板进行定向修复, 可以实现特定位点的精确插入、缺失或者碱基置换。而非同源末端连接不依赖模板直接连接DNA两端, 可以在DSB突变位点有效产生不同长度片段的插入或缺失, 通常这种修复快速但不精确, 而主要利用这类非同源末端连接对定点诱导的DSB的非精准修复, 便是基因编辑实现的主要途径。实现体内特定遗传位点的DSB的精准诱导, 便成为了实现对体内遗传信息进行改变的所谓“基因编辑”的关键。先后有多种基因编辑技术得到报道, 而自2010年以来, 对酿脓链球菌(Streptococcus pyogenes)等细菌和古细菌中的一类抵御病毒和质粒DNA入侵的核酸内切酶的适应性免疫系统, 即成簇规律间隔的短回文重复序列(Clustered Regularly Interspersed Short Palindromic Repeats, CRISPR)及相关蛋白(CRISPR associated protein, Cas)组成的CRISPR-Cas系统[2, 3], 其设计特定核苷酸序列的识别和结合的便利性, 内切酶效率的稳定性, 使其成为了当下基因编辑的主流技术, 已成功实现了对人类特定细胞培养物[3], 以及酵母[4]、细菌[5]、果蝇[6]、小鼠[7]和斑马鱼[8]等生物的在体基因编辑应用。
1.2 基因编辑技术与农业生物应用
起初CRISPR-Cas系统主要应用于构建各种生物基因生理功能和机制的在体研究模型[3]。随着以基因编辑技术构建的生物品系呈现出的各类特征性表型, 基因编辑技术很快成为生物育种的应用研究和探讨热点。不同于既往利用转基因技术, 利用基因编辑技术改良的生物, 将不会有外源功能基因向目标物种基因组的导入, 例如转基因水稻中转入来自苏云金杆菌的抗虫基因——伴胞晶体毒素[5], 以及来自土壤细菌中的抗杀虫剂基因—— 5-烯醇丙酮莽草酸-3-磷酸合酶等等[9], 这些外源基因产物的潜在外源蛋白及毒性效应风险, 较易引发公众和毒理学工作者所关注。
过往对农业生物性状的调控网络解析的研究, 许多生产性状的关键调控基因的甄别已有了长足的进展。对于农业生物性状改良的优势传统育种手段难以实现的农业生物经济、品质性状的改良, 精准、快速的基因编辑技术带来了如虎添翼的新机遇。例如利用基因编辑技术改变奶牛的POLLED位点, 导致无角奶牛的品系建立, 从而减少对挤奶等操作人员和奶牛相互间角斗的损伤[10]; 利用基因编辑技术分别实现的对牛CD46、猪CD163的改变, 可使牛的牛病毒性腹泻病毒(Bovine viral diarrhea, BVDV)、猪的猪繁殖与呼吸综合征(Porcine reproductive and respiratory syndrome, PRRS)的受体分子发生功能改变或缺失, 从而产生对牛病毒性腹泻病毒(BDDV)、或猪繁殖与呼吸综合征病毒(PRRS)感染(亦称为“蓝耳病”)的抵抗能力[11, 12]。一些中南美洲的土生肉牛的一类催乳素受体(Prolactin receptor, PRLR)位点, 具有一种独特的单核苷酸多态性 (Single nucleotide polymorphism, SNP)变异, 使这些牛的毛发较短, 较为耐热。而另一方面, 选育自北欧的奶牛、肉牛品种虽具有较强的产乳和生长优势, 但在当前气候变暖情况下, 夏季生长和产奶出现较大损失。若采用传统选育方式, 把这些热带和温带牛进行交配, 向北欧品种导入热带品种的PRLR耐热的SNP位点, 将需要较长时间和代数将这些优势性状适当再整合和再选育。而借助基因编辑技术, 成功实现在北欧品系中人工精准导入热带品种的PRLR位点样的突变, 快速实现其耐高温的性状改良, 同时基本保持温带品种的生长和产奶性状[13, 14]。在猪养殖过程中, 常采用公猪阉割操作, 因为去势公猪在养殖过程中便于管理、改善肉质、促进生长, 但其过程的残忍常被人诟病。利用基因编辑使性腺发育的重要基因Kisspeptin 1 (KISS1)发生改变, 获得了性腺发育迟缓、温顺和生长较快的公猪, 从而改善了动物福利和养殖工人的操作[15]。牛、羊奶中的β-乳球蛋白对许多人, 尤其是对婴儿来说是主要的过敏原, 易在人群的引发过敏反应。最近, 中外学者利用基因编辑技术, 特异性地改变牛、羊中的β-乳球蛋白(β-lactoglobulin, BLG)编码基因的结构, 可获得无、或β-乳球蛋白低含量的牛、羊奶[16—18]。在作物育种方面, 则利用基因编辑技术针对抗杀草剂、抗病虫害、耐气候变化引起的环境胁迫, 以及增强作物生长与富含有利于人类的营养物质等方面开展研究[19], 如对水稻Tubulin A2编码基因OsTubA2的单氨基酸转换导致的对二硝基苯胺类除草剂的抗性[20]、编码水稻半卷叶1, 2的基因Semi-Rolled Leaf1,2 (OsSRL1,2)的基因定点编辑增强水稻耐旱能力[21]、编码水稻应激调控蛋白22 (Response Regulator 22)的OsRR22基因定点编辑增强水稻耐盐能力[22], 以及对西红柿谷氨酸脱羧酶2和3蛋白(Glutamate decarboxylase 2, and 3, GAD2, 3)的编码基因SLGAD2和SLGAD3的定点编辑, 在西红柿中提高有助于高血压、高血脂症的缓解的γ-氨基丁酸(Gamma-aminobutyricacid, GABA)含量[23]。
作为全球基因编辑辅助育种的应用大国, 美国自2009年就已批准借助Talens技术辅助改良的大豆商业化应用, 目前各类基因编辑育种的各类农作物种植面积约为1.85亿英亩, 约占目前全球基因编辑作物的种植面积的39%[24]。早在2020年, 也已批准用于肉食和治疗用途的“半乳糖苷安全猪”(GalSafe pigs)的订购销售, 用以预防蜱虫叮咬咬引起的对红肉(猪牛羊)和乳制品的食物过敏症人群, 即对所谓α-半乳糖综合征(Alpha-gal Syndrome, AGS)防治[25]。我国农业农村部于2022年制定公布了《农业用基因编辑植物安全评价指南(试行)》之后, 对农业用基因编辑植物的安全评价进入正轨, 已自2023年4月28日始, 目前已发布了3批共5种基因编辑大豆、小麦品种的生物安全证书(生产应用)[26—28]。目前, 我国尚未发布有关农用基因编辑动物的安全评价指南, 尚无基因编辑动物通过生物安全论证。
2. 基因编辑技术在鱼类育种中的应用
和传统选育, 甚至新近发展的分子标记辅助育种、基因组指导选育、杂交育种等方法比较, 在淡水鱼类的基因编辑操作较易实现, 基因编辑技术对特定基因进行定向精准编辑, 实现对鱼类生产性状的精准、显著改良, 甚至是颠覆性的创新改良。我国淡水鱼类养殖产量居全球第一, 近期对基因编辑技术在鱼类性状改良的论文作者统计, 中国学者发表的文章占全球发文量的最高份额[29], 这显示了我国学者在该领域的国际领先地位。
斑马鱼作为鲤科模式鱼类, 借助基因编辑技术, 已开展了大量的基因功能研究, 涉及有关生产性状包括: 生长[8, 30—32]、抗病[33, 34]、耐低氧[35, 36]、饲料转化率提升[31]和倍性操作[37]等。由于许多鱼类生长存在着性别异形, 因此, 养殖具有生长优势的单性别鱼类群体, 将带来养殖产量的增长, 为此利用模式鱼类斑马鱼开展的鱼类性别分化与调控的研究, 既具重要生物学理论意义, 也具有显著的产业应用价值[38—45]。鲤科养殖鱼类对消费者产生的一个较大困扰就是肌间刺的存在, 自20世纪60年代以来, 各国研究者曾利用人工选育、杂交、雌核发育、多倍体选育以及分子标记辅助选育等育种手段, 从未在鲤中获得肌间刺减少超过50%的效果[46], 而高泽霞团队借助斑马鱼单细胞测序分析和大量候选基因的基因编辑敲除的表型观察, 终于通过敲除RUNX family transcription factor 2b (runx2b)基因, 成功地获得了无肌间刺的鲤科鱼类[47], 这一成功案例显示了相对于传统选育技术, 基因编辑技术的对鱼类品质性状改良具有难以比拟的精准、快速效果。
基因编辑技术应用于养殖鱼类的性状改良也吸引了众多学者的目光, 自2014年以来, 针对养殖鱼类的基因编辑改良开始见诸报道[48, 49]。迄今为止, 由于鱼类繁育和鱼卵硬壳等问题, 多数养殖鱼类基因编辑的研究, 主要以淡水中繁育的鲤科、慈鲷科(罗非鱼)和鲑科鱼类中开展, 针对的主要生产性状是性别与繁育、体色、生长与抗病[50]。我国学者则曾经在黄颡鱼、半滑舌鳎、罗非鱼、异育银鲫、泥鳅、鲤、南方鲇、团头鲂、鲫和黄鳝等养殖鱼类开展基因编辑研究, 主要针对的性状包括生长、性别调控、肌间刺等性状改良,已获得多个稳定遗传的性状改良品系, 其对生产性状的改良效果与传统选育效果相比极为显著(表 1)。
表 1 我国利用基因编辑技术开展的养殖鱼类生产性状改良研究报道Table 1. Improved traits of farmed fish utilizing genome editing in China养殖对象Farmed fish 突变基因Mutant gene 性状与改良效果Improved trait 年度Year 参考文献Reference 黄颡鱼
Pelteobagrus fulvidracomstnb F1代生长至210日龄, 体重增加137% 2011, 2014, 2020 [51—53] 尼罗罗非鱼
Oreochromis niloticusamhy, mstnb F0代突变体显示amhy是尼罗罗非鱼雄性决定的必要基因
F2代缺失mstnb纯合子雄鱼相较对照组体重增加49.45%2015, 2023 [54, 55] 半滑舌鳎
Cynoglossus semilaevisdmrt1 F0代突变体显示dmrt1是雄性决定基因 2017 [56] 团头鲂
Megalobrama amblycephalamstna, mstnb F0代杂合子分析, mstna突变体体重增加7.98%; mstnb突变体体重增加6.79% 2020 [57] 异育银鲫
Carassius gibeliopik3r1 F2代杂合突变品系分析, 年度生长提升10%以上, 饲料转化率提升10%左右 2021 [58] 泥鳅
Misgurnus anguillicaudatusmstn F0代突变体促进生长可达34.9% 2021 [59] 异育银鲫Carassius gibelio foxl2, gsdf,
cyp19a1a利用各类突变体, 研究了多倍体中各基因的不同同源基因亚型在繁育调控中的功能分化 2021, 2022 [60, 61] 黄河鲤Cyprinus carpio cyp17a1 F2代纯合子分析, 全雌单性别黄河鲤群体较同塘混养群体年度生长提升20%以上 2022 [62] 南方鲇Silurus meridionalis amhr2y F0代突变体证实amhr2y具有雄性决定功能 2022 [63] 团头鲂
Megalobrama amblycephalarunx2b F2代纯合子分析, 完全消除纯合突变的团头鲂肌间骨 2023 [64] 异育银鲫Carassius gibelio runx2b 完全敲除的纯合突变的异育银鲫肌间骨 2023 [65] 鲫Carassius auratus bmp6 F2代纯合子分析, 完全消除纯合突变的鲫肌间骨 2023 [46] 黄鳝Monopterus albus cyp19a1a, foxl2 F0代突变体显示cyp19a1a, foxl2基因对卵巢发育的调控影响 2023 [66] 3. 基因编辑育种应用的生物安全风险
鉴定特定性状调控的关键基因位点需要前期大量基础研究, 基因编辑技术通过对性状调节的关键基因某个或多个位点诱发原位DNA突变, 这些突变通过生殖细胞遗传到下一代。对获得的下一代个体进行突变基因型筛选并对突变表型进行评估, 从而获得对生产性状有利的稳定品系。因此, 基因编辑技术应用于生物育种, 获得的依赖人工诱导物种的遗传物质改变, 即携带了可遗传的“人工编辑位点”, 这些位点并非自然发生的遗传变异或称“天然变异位点”。这也是基因编辑辅助育种和传统选育育种的最本质的区别。传统育种凭借对大量育种对象的表型观察和筛选, 通过对具有产业优势的优良表型性状个体收集、选择、传代, 将这些对生物群体中出现的、具有一定优化价值的性状关联基因中出现的“天然变异位点”进行筛选, 通过繁育和传代, 使目标性状的调控网络及其关联位点所出现的 “天然变异位点”, 在养殖群体中获得不断地、尽可能多地富集, 使目标性状得以持续优化。传统的选育是一个漫长的过程, 它包含优良的“天然变异位点”的等待、筛选、交配、培育和传代的过程。
目前国际上将基因编辑的遗传改良品种分为三大类别: (1)单个、数个碱基缺失; (2)设计并定向取代碱基, 导致新功能蛋白生成; (3)定点发生外源基因整合(不同于转基因技术常会发生外源DNA的随机性插入)。目前, 基因编辑技术目前多采用的是CRISPR/Cas9系统, 其改良生物一般是产生的第(1)类改良品种, 不产生外源DNA的插入。而对基因编辑生物的生物安全管理最主要的关注点: a. 是否存在外源DNA的插入和留存; b. 发生序列改变后形成的新编码的蛋白的毒性毒理学效应或新生过敏原效应; c. 基因编辑过程中是否存在意外的脱靶效应, 从而产生了非靶标位点的遗传变异, 导致未曾预料的新编码蛋白的安全效应未获充分测评。
3.1 食品安全风险
基因编辑育种导致的生物安全风险, 一般可归为食品安全风险和生态安全风险。而食品安全风险, 则主要针对基因编辑育种获得的新品种食材, 包括作为兽用饲料, 或是作为人类食品用途, 所可能带来的风险。对人类基因编辑技术辅助育种的生物材料尽管一般不存在外源基因编码的蛋白, 但因原有DNA编码区发生改变, 导致DNA编码氨基酸的密码序列移码, 形成与原编码蛋白不一样的“新蛋白”。尽管这类所谓的“新蛋白”, 并非与任何已知蛋白一样的肽链, 即并不是“外源物种”的已知蛋白, 但毕竟是该改良物种体内产生的一种原来不存在的“新蛋白”类物质。而这类因为基因编辑而导致的“新蛋白”物质, 是否会对食物链中的不同层级的消费者、甚至人类的食用后产生毒理学效应、过敏原效应? 这些评价工作过去主要通过细胞培养物、动物体的各类体外、体内试验进行评价, 例如慢性的、急性的各类毒性试验, 对“新蛋白”的潜在的风险进行评价[67]。同时, 也有国内外学者和国际组织, 开始构建大型数据库, 集蛋白同源序列与结构的数据收集, 运用大数据搜索和智能计算, 以非实验的手段, 先行对“新蛋白”进行大规模序列同源性和结构比对, 从而预测其潜在食品安全风险, 再进行必要的、针对性的食品安全测评, 相信这些技术的应用, 将对未来基因编辑动植物品种的食品安全论证提供重要的帮助[68, 69]。
3.2 生态安全风险
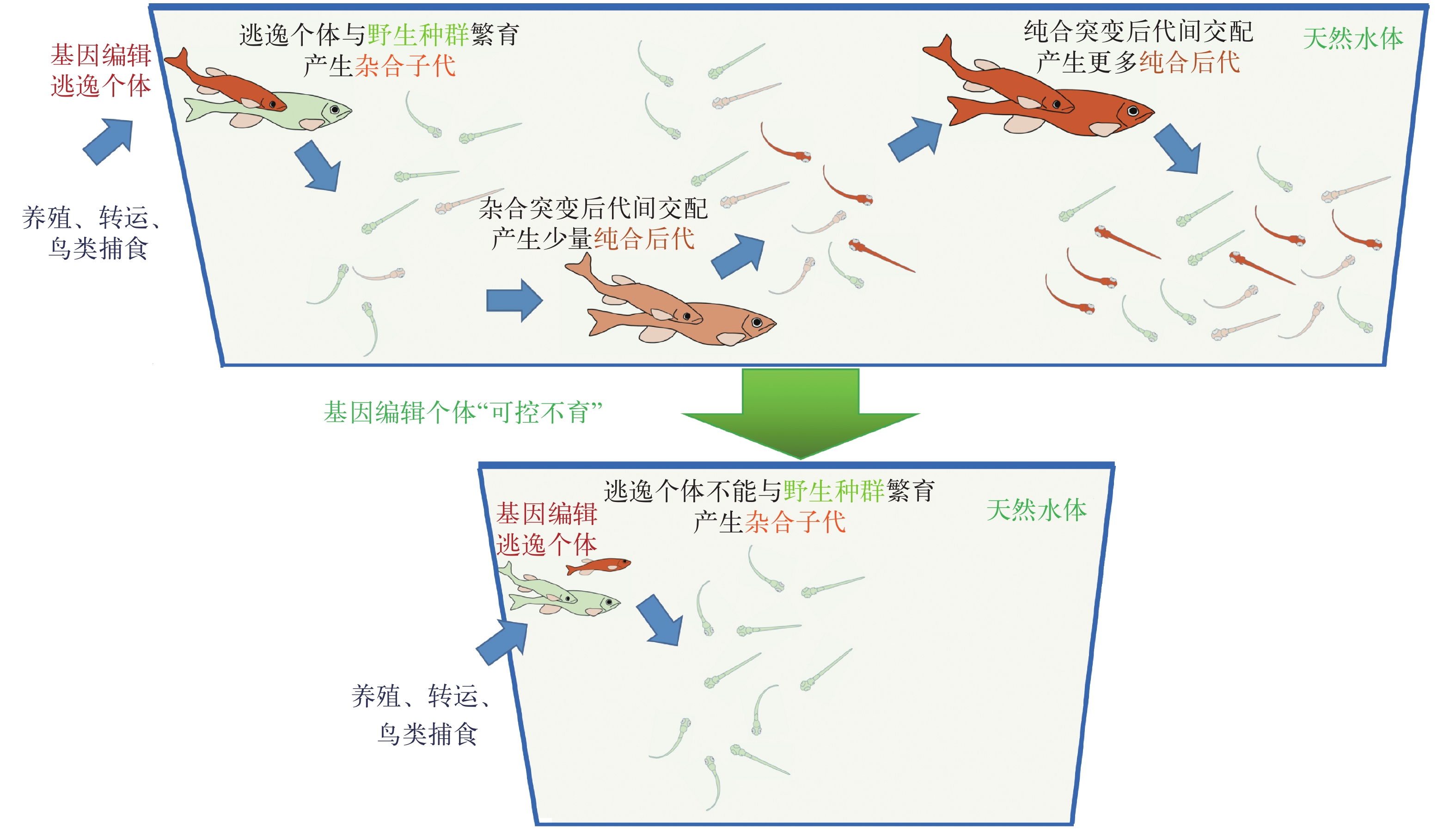
实际上, 任何遗传育种过程包含基因编辑辅助育种, 均会导致改良品种与野外种群间出现遗传差异。经多代遗传选育的大多数水产养殖品种, 和野外种群的存在遗传差异。鱼类对生产过程的适应所进行的驯化过程, 常伴随着遗传多样性的减少[1, 70]。例如, 经2—3代优选自交的草鱼养殖群体, 检测到某些特定位点的杂合子缺失, 相对于野生群体的遗传多样性下降[70]。源自挪威的野生种群, 经过12代驯化与选育的大西洋鲑, 与来自挪威、加拿大等的4个野外区域采集的大西洋鲑DNA样本, 进行约5000个SNP标记的比较分析时, 一些特定基因组DNA区域和44个SNP多态位点仅在选育品种中出现, 显示其成为与该驯化选育品种的 “遗传标签”, 这些位点主要涉及促进生长和延缓繁育的关联信号分子[71, 72]。Karlsson等[73]在挪威109条鲑鱼洄游河流进行野外鱼类采集, 发现在具有驯化培育养殖品种“遗传标签”的样本数量, 可以占这些河流中采集的野外个体的0.0%至42.4%, 平均值为6.4%。这表明驯化培育养殖品种可在养殖过程中出现“逃逸”, 进入野生水体。若“逃逸”个体与野生种群在野外水体中进行繁育, 便可产生遗传物质交流, 导致驯化培育的“遗传标签”在野生种群中发生遗传漂移, 对获得并携带该类“遗传标签”的野生个体产生一定的生理功能影响。考虑到养殖品种的驯化和选育, 常导致促进生长和延迟繁育等现象, 使养殖对象的“逃逸者”具有生长快、身体尺寸大的优势, 从而在野外群体易获得较多繁育机会, 但其较为延迟的繁育性能, 却又可能延滞和影响后续种群繁衍, 导致种群数量的下降, 造成种群生态结构和数量劣势, 影响原有生态系统[74, 75]。另外, 高密度、低应激、抗病、体色等经济性状也常常是养殖对象驯化与改良的目标, 由此而产生的“遗传标签”向野生种群的漂移, 对自然资源的维护和生态系统的影响, 一时也难以评估。因此, 即便是经传统的驯化选育的养殖品种“逃逸者”, 都可能通过因其 “遗传标签”在野生群体中的渐渗, 对受纳水体的生态系统产生难以预料的生态影响。
水产养殖对象的生存环境与海淡水系统密切联通, 使水产生物在养殖过程中发生的“逃逸者”, 相对于陆生种养动植物的“逃逸者”具有更为隐蔽、更远、更广的迁移距离和尺度[76], 而且对于水产养殖过程所产生的“逃逸者”, 也难以实现有效、快速的鉴别、回捕[77]。导致养殖“逃逸者”出现的原因常包括洪水飓风对养殖设施的损坏、养殖鱼类自养殖水体逃逸、活体转运、受精卵漂移、鸟类捕食等。据报道, 2017年中国因台风和洪水影响养殖面积120万hm2, 造成养殖产量损失约60万吨, 多数发生在中国南方水域[78]。作为水产养殖大国, 我国学者虽曾有报道逃逸的养殖鱼群体在野外被检测到[79], 但总体而言, 我国学者针对养殖驯化鱼群在野生鱼类群体中的出现数量、比例和影响, 研究报道很少。相对而言, 欧美学者则在此方面进行了更多研究。地中海区域是世界海水鱼类网箱养殖的热点地区之一, 在2015年前后, 常年约有2万网箱养殖着400—500亿尾各类鱼类, 据估计在2013—2015年, 约发生765万尾成年和幼鱼“逃逸”, 以至于在某些局部区域的鱼类捕获物中20%—70%来自“逃逸”鱼[80]。正因为养殖场所的“逃逸者”, 有可能带来对野生种群原有遗传构成的影响。自2009年开始, 挪威水产管理部门颁布与执行了日益严格的海水养殖的管理规定(NYTEK NS 9415), 制定了防止“逃逸”的设施建设要求和管理措施。自规定颁布执行以来, 挪威报告的“逃逸”鱼类自2006年的120万尾, 已降至2015年的20万尾以下[81]。但毕竟由于海洋灾害、交通运输事故等因素复杂多变, “逃逸现象”依然难以彻底杜绝, 有学者依然悲观地认为只有更为严格地实行养殖方式的管理, 进一步显著地降低“逃逸现象”, 或是采用“不育”的对象进行养殖, 才能杜绝未来由养殖物种向野生物种“遗传渐渗”, 使选育鱼类的养殖导致的生态风险威胁得到消除[82]。
相较于鱼类传统选育, 基因编辑技术辅助的水产生物育种是近十年来出现的新事物。由于基因编辑技术定向导致的遗传突变位点, 是人为的目标导向操作而形成的“人工位点”。如果养殖活动中的携带“人工编辑位点”的逃逸个体, 具有与野生种群在野外环境中发生繁育活动的能力, 产生遗传物质交流, 也将导致“人工编辑位点”在野生种群中遗传漂移, 如同前人所述的关于养殖对象的选育关联 “遗传标签”一样。对养殖鱼类开展基因编辑辅助育种的报道(表 1), 如性别转换、肌间刺的消除等, 表明这些“人工编辑位点”是目标性状调控的关键位点, 不同于传统选育呈现的调控关联基因的“量变垒进”多代选育进程, 基因编辑则是在1、2代繁育中, 精准地对性状关键调控基因实现“质变跃进”选育进程。因此, 其“逃逸者”若对野生对象产生遗传渐渗, 将对获得“人工编辑位点”的野生个体产生更为显著的生理功能影响, 从而可能影响野生种群原有的生态功能或其“生态位”, 对水体中的野外种群资源维护, 甚至水生态系统产生难以预期的生态安全风险。
4. 基因编辑育种生物安全防控
基因编辑辅助育种由于产生了品种的遗传结构的变化, 一般在各国都是归为了遗传改良生物(Genetically Modified Organism, GMO), 加以生物安全管理。但相较于引入了“外源功能基因”的转基因生物, 目前基因编辑育种产物, 一般较易得到管理者和消费者认同。目前不同的国家在基因编辑培育的市场化应用管理方面存在不同观点。一种观点认为, 基因编辑技术导致的“人工编辑位点”, 由于变异较小, 许多“突变”类似于可在天然条件下自然产生的点突变、或是遗传多态性, 与物种的自然演变较为类似。而且突变选育按照人类生产需求而进行的性状“改良”, 多会使养殖对象, 在经过长期演化形成的对野生环境适应生理稳态, 发生不利改变。尽管, 这些“突变”可能在特定养殖条件下, 使改良的生物得到生长加速、繁育加速, 或是抵抗某种养殖条件下更易发生的疾病等等。因此, “逃逸者”往往并不益于在野生环境中形成有影响力的优势群落。反而, 在天然状态下, 这些 “人工编辑位点”携带者最终较易被淘汰, 难以对野生群落产生显著而持续的影响, “人工编辑位点”最终在野外群体得以清除。目前, 日本、阿根廷等国已采用这类较为宽松的“准予基因编辑育种试用”的方式。根据报道, 美国水产育种公司AquaBounty的mstn基因编辑的尼罗罗非鱼(编号FLT01)曾于2018年率先获批在阿根廷上市; 2021年, 日本批准了富含γ-氨基丁酸(γ-AminobutyricAcid, 简称GABA)的基因编辑番茄, 以及肌肉组织增加的双斑东方鲀(Tiger puffer)和真鲷(Red sea bream)上市, 这些基因编辑技术改良的鱼类都使其出肉率或生长速度出现了超过20%的提升, 饲料转化率提升14%以上[83, 84]。尽管如此, 在巴西等国, 如果在基因编辑辅助培育的动物发现含有已知外源DNA序列, 如质粒序列, 则将归为转基因类别的遗传改良生物加以管理。例如在巴西, 原本作为基因编辑肉牛批准商业化使用, 后因被美国食品药品监督局检测出其中含有转基因质粒的DNA序列, 而被巴西管理机构取消商业化许可[85]。
4.1 食品安全评价
基因编辑常导致生物体内出现提前终止的“截断蛋白”或是“移码新蛋白”, 由这些基因编辑而导致的这类新形成的编辑蛋白, 是否会形成具有某种功能性的蛋白, 或其类似的同源性蛋白? 需要进行认真地评估和测试。例如, “新蛋白”在编辑生物体内表达组织和表达水平、在活体或在存储和食物加工后的蛋白稳定性及代谢物水平; 蛋白序列在已知生物信息库中开展比对, 查明其是否具有显著同源性的蛋白或是功能基团, 再判断其可能具有的生物活性、生物毒性、免疫原性和致敏原性等功能; 然后对新蛋白的合成产物开展针对性的体内、外相关实验研究、研究验证。国内外相关研究学者和管理机构曾对此, 提出和建议了多种检验流程, 但目前还尚未形成国际统一规范[86]。在我国, 对于基因编辑鱼体, 需要在《中华人民共和国食品安全法实施条例》的规范管理下, 由国家管理部门指定的第三方资质机构, 经过论证的流程, 开展生理、毒理实验, 以及相关食品安全动物试验, 出具测评报告。例如, 通过委托具有水产品食品安全管理资质的机构——农业部农产品质监检测中心, 已初步完成了对基因编辑全雌黄河鲤的食用安全论证[87]。
4.2 生态安全防控与育性控制
更多的欧美和中国学者则持更为谨慎的观点。认为毕竟基因编辑技术定向导致的遗传突变位点, 是非天然产生的“人工编辑位点”, 如果水产养殖活动难以避免的“逃逸者”, 与野生种群个体发生交配, 会产生不同于天然形成的、人工选育关联 “遗传标签”漂移, 因此, 许多学者针对这类可能产生“人工编辑位点”的“逃逸者”, 在受纳水体中与野生种群进行繁育, 甚至最终形成纯合“人工编辑位点”的“突变”群体。因此, 学者们非常关注其产生对野生种群, 甚至水体生物群落的影响与评价(图 1)。许多学者借助既往对外来入侵物种的生态风险评价与管理经验, 提出了遗传改良生物的生态安全评价与管理模式。将基因编辑改良物种“逃逸者”与引入“外来物种”对等, 重点针对编辑改良物种“逃逸者”在潜在受纳水体中的适应能力、繁殖能力和扩散能力等三个方面进行考评, 全面考量各方面生态风险, 这些可以细分为编辑鱼类的生存适温范围、盐度适应范围、适氧范围、耐受其他常见水体中的污染物范围、易感病原范围、食性与捕食能力、个体产卵量、胚胎与仔稚鱼发育适应能力、开口饵料满足的难以程度、繁育代时长短、运动与自然传播能力以及人为传播能力等等方面; 建议予以关注的方面也还包括携带“人工编辑位点”的“逃逸者”, 所具有的特征性改变的生理性状, 对该物种及其近缘物种的野生群体带来的生态种群结构和功能发挥的潜在影响, 甚而对受纳水体整体生态系统功能发挥的影响。当然, 也有学者建议对编辑改良物种所导致的优势, 包括对动物和人类福利的正向影响、对资源可持续利用和人们营养改善等正向影响等因素, 均需一一赋分, 进行全面评估[85, 88, 89]。可以设想, 这些非天然的“人工编辑位点”一旦融入在野生群体, 也的确难以短时在野生环境中得以完全清除, 而其与野生同一物种个体间交配、不同物种间杂交等事件的发生, 所带来的物种生理改变和其生态位功能的影响较难以穷尽地对其的生态风险进行客观评价。因此, 许多学者认为, 应本着尽量避免“逃逸者”与野生种群发生繁育, 从而产生遗传漂移为原则。那么就希望做到基因编辑种质的“可控不育”, 即用于养殖、运输和商业化过程等的养殖对象, 应做到基因编辑的养殖对象“不育”, 而在生物安全严格控制的制种场所, 能可控地恢复育性, 以保证改良种质的供应[88, 90—94]。欧共体针对这方面的管理流程, 是按照编辑品种的“可育” “不可育”的不同类别, 提交不同的环境影响评价报告, 其中“可育”品种类别, 需要针对“逃逸者”对受纳水体的潜在影响, 提供更多的、更为全面的环境影响评价报告[75]。
对于携带有“人工编辑位点”水产动物在水产养殖过程中出现的逃逸, 及其在野外鱼类中产生遗传渐渗的生态风险的担忧, 学者普遍认为“可控不育”技术, 是养殖鱼类养殖活动产生生态风险的最佳控制手段[82, 88, 90—94]。近年来, 有关鱼类可控不育, 或是可控育性、诱导育性、生殖开关等的研究成为了鱼类育种共性技术研究的热点。迄今为止, 通过对鱼类遗传操作研究, 有可能实现 “可控不育”的策略大约分为3种: (1)种间杂交技术, 导致的杂交品种不同亲本, 形成非整倍体基因组的子代, 将杂交子代作为养殖对象, 这些养殖对象便能实现“不育”。例如朱作言院士与刘少军院士团队合作获得的三倍体生长激素转基因鲤杂交后代不育[95], 以及刘少军院士团队构建的多个利用的鲤、鲫杂交后代“湘云鲫”, 将性状改良的鲤、鲫编辑品系进行杂交, 则可能实现具备编辑位点改良了性状的杂交后代不育[96]。但此方法一般需要依赖杂交种对性状表现无不良影响, 且仅限于特定的种间杂交可行品种或对象; (2)同种或异种鱼的不同倍性利用特殊交配方式导致子代因基因组异倍性而“不育”; 这方面的代表性案例是近期桂建芳院士团队研发的合成新双三倍体银鲫的方法, 该双三倍体“维持系”可以“无减数分裂融合”生殖方式, 与鲤科鱼类雄性个体进行繁殖, 产生不育的异源七倍体, 发展出一条高效创造不育的异源多倍体的技术路线[97, 98]; (3)基因编辑导致的繁育缺陷, 辅以有效繁育的挽救手段, 使“繁育缺陷”能得以有效挽救, 从而以其为亲本, 实现子代“可控不育”。该方法将利用基因编辑改变育性的同种个体间实现育性的某种挽救, 一旦实现, 则可以不需要考虑异倍性和杂种个体对特征改良的性状的影响, 具有较强的普适性。只是基因编辑技术产生“突变体”常能使育性完全丧失, 但再有效地恢复受损育性的配套策略, 一时难以实现, 故还未有成功的报道案例。国内外学者, 前期多针对原始生殖细胞(Primordial germ cells, PGC)早期发育容易被基因敲除破坏来着手。例如, 敲降dead end (dnd)基因可导致PGC缺失, 造成鱼类因无生殖细胞而不育。这些技术包括: 在养殖鱼中使用吗啉基寡核苷酸(Morpholino, MO)显微注射, 敲降DND蛋白, 破坏鱼类PGC; 也有报道是采用酵母Gal4重组蛋白(含有一种DNA序列结合基团BD和RNA转录激活基团AD)/Gal4 BD的结合序列UAS (Upstream activation sequence, UAS)体系, 构建转基因斑马鱼品系, 通过交配构建诱导dnd反义负向序列表达, 获得利用转基因斑马鱼品系, 实现交配后的子代, 因生殖细胞缺失而不育斑马鱼体系[90, 99]。这些方法后续尚未见在养殖鱼类的试验体系中实现可控繁育的成功报道。一般认为, 若采用每次向受精卵进行dnd MO呈递, 工作量之大, 难以在产业应用中便捷实现; 而构建GAL4/UAS转基因体系, 也将因属于含有酵母GAL等外源蛋白的转基因生物, 且在鱼类组织中的诱导效率的稳定性问题, 需要进一步的后续研究。还有报道, 通过基因编辑构建dnd敲除的大西洋鲑, 采用回补dnd RNA呈递的办法挽救早期鱼卵, 显示可以使大西洋鲑幼鱼中出现生殖细胞, 即对早期生殖性状即受损的个体, 出现了有希望的育性恢复[100]。但后续未见成功实现繁殖的报道, 而且, 相信此种策略, 即便能恢复, 其操作手续也较复杂, 成本较高, 整个恢复过程难以控制。同样, 依据致繁育受损再恢复的思路, 近期我们团队, 另辟蹊径, 针对成年阶段育性障碍进行恢复。我们利用对促黄体素β (Luteinizing hormone subunit β, lhβ)的敲除, 将使雌性斑马鱼卵子发育受阻于IV期卵泡发育期[101, 102], 通过成功研发其有效的针对性助育剂, 成功挽救了lhβ敲除的雌性斑马鱼的繁育能力[103]。近期, 我们借助已成功构建cyp17a1基因编辑全雌黄河鲤的研究基础[62], 进一步敲除黄河鲤的lhβ基因, 在特殊的助育剂辅助下, 实现了采自cyp17a1–/–; lhβ–/–伪雄黄河鲤父本的精子与lhβ–/–不育雌性黄河鲤母本的卵子, 在体外的人工受精, 批量获得了不育、全雌黄河鲤(cyp17a1+/–; lhβ–/–), 而且 cyp17a1杂合突变、lhβ纯合敲除突变(cyp17a1+/–; lhβ–/–)的黄河鲤的全雌及不育的表型已得到证实(未发表结果), 从而以全雌+雌性不育的二步法, 实现了黄河鲤的不育, 再通过特殊的助育剂的采用, 实现了育性的挽救, 达到了育性可控的目标。
5. 鱼类基因编辑育种的应用展望
鱼类基因编辑技术辅助性状改良的研究, 在鲤科模式鱼类斑马鱼中已体现出了包括生长、抗病和品质改良的显著效果, 随后也在鲤科鱼类、罗非鱼和大西洋鲑等主要鱼类养殖对象中, 对促进生长、性别控制、无肌间刺和育性可控方面取得了可喜的成功。目前, 除了在日本和阿根廷, 基因编辑改良鱼尚未有获批上市, 但相信在不久的未来, 基于养殖鱼类“可控不育”研究的进一步完善和推广, 将使鱼类基因编辑技术辅助性状改良的产业化应用得到认可。当然, 依托对鱼类基因组、表型组研究的进步, 在鱼类性状调控机制的知识理论的增强, 借助分子标记辅助育种、全基因组辅助育种等技术, 利用群体中自然发生的“优良等位变异”, 进行鱼类的选育效率也会得到不断的提高, 考虑到公众对自然发生“变异位点”的选育品种具有较高的认同感, 一般认为, 基因编辑辅助进行的鱼类育种工作, 应主要关注对重要经济性状具有“跨越性”“飞跃性”“颠覆性”的改良应用, 首先选择难以通过对自然发生的“优良等位变异”选育改良的性状。
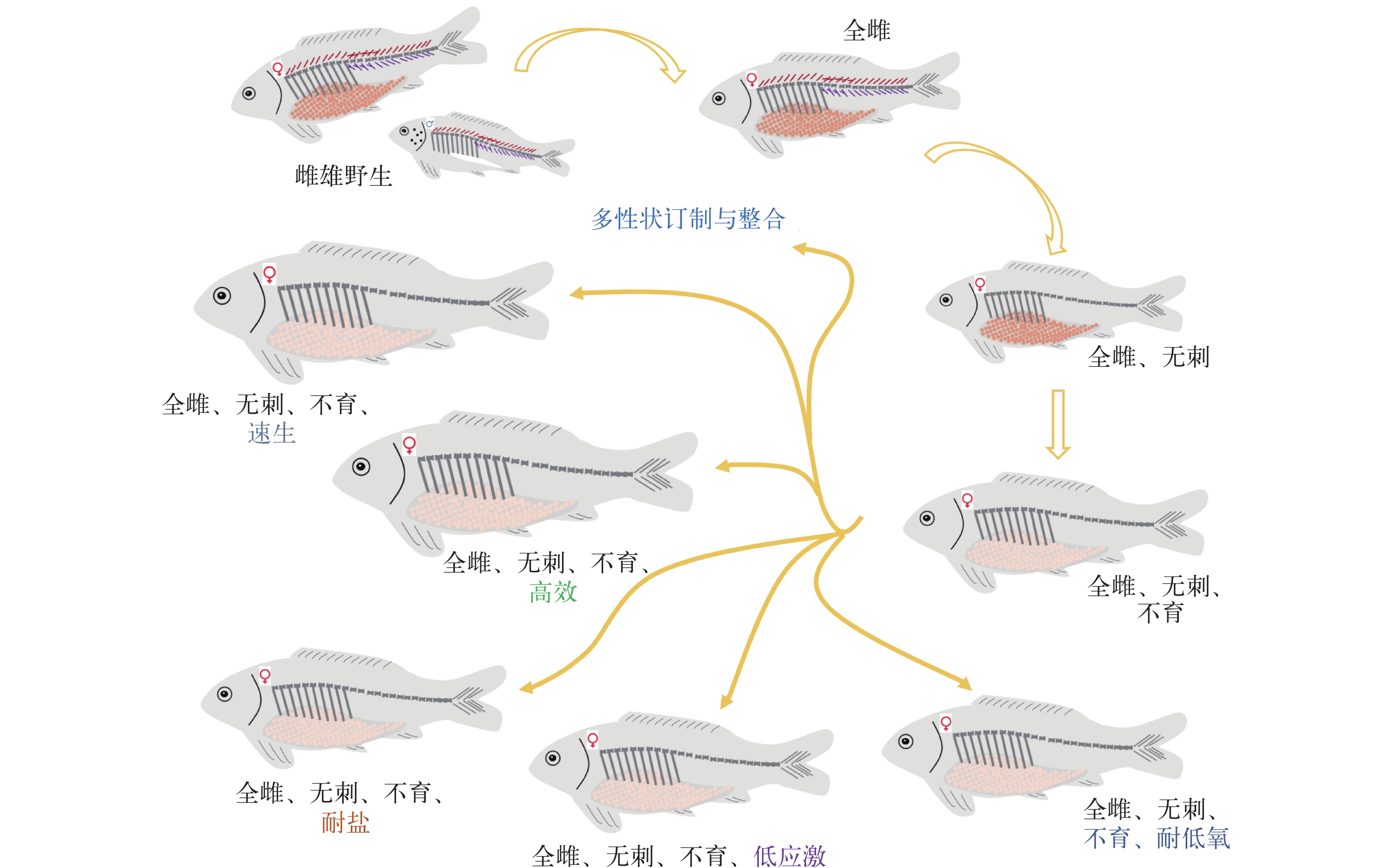
我国常见的大宗养殖鱼类多属鲤科鱼类, 由于适应性强, 食性广, 自古以来便是我国主养对象, 其饮食风味也深入人心。2022年, 我国鲤科养殖鱼类产量占淡水养殖鱼类总产量的74.75%[99]。利用鲤科模式鱼类斑马鱼、鲫、鲤、团头鲂等所开展的研究, 证实了上述调控机制在鲤科鱼类中具有高度的保守性, 因此, 相信这些技术也可以适用于其他鲤科鱼类, 如青鱼、草鱼、鲢、鳙、鲫、鲂等。鲤科鱼类具有较多的肌间刺, 将已有杂交鲤鲫、异倍体不育鲫、基因编辑全雌且增产不育鲤等成果, 与敲除控制肌间刺等性状改良结合, 将可以获得无肌间刺的杂交不育鲤鲫、异倍体不育鲫和全雌增产不育黄河鲤群体, 既做到让消费者不再担心“卡脖子”的鱼刺, 也消除人们对基因编辑鱼类生态风险的担忧。
当然, 已获得初步成功的养殖鱼类的各类“可控不育”技术尚在起步阶段, 未来在产业化应用方向上, 在下列许多方面尚需进一步完善和研发: (1)进一步开展鱼类育性调控机制研究, 开发更多的育性控制与挽救方案。例如研发更多获得雌性鱼类不育缺陷及育性挽救的可行、便利方案, 提升繁育缺陷的挽救效率, 降低挽救育性操作的成本, 提升产业应用收益; 同时, 探索雄性养殖鱼类的不育及挽救体系研究, 拓宽“可控不育”的应用范围; 应对基因编辑鱼类育种的生物安全论证, 为“可控不育”养殖鱼产业化构建示范基地, 实现规模化应用。(2)创新自主知识产权的鱼类基因编辑底盘技术, 在此基础上开展鱼类性状改良的产业化的应用研究。(3)进一步开展养殖鱼类经济性状调控关键靶标分子甄别的基础研究。在“可控不育”鱼类基础上, 整合无肌间刺、抗病、抗逆(低氧、高盐、高温)、高饲料转化率等性状改良鲤科养殖鱼类系列、多性状整合改良品系系列, 实现以基因编辑技术辅助的精准定制化品种培育(图 2), 推动水产种业科技进入以企业为主体的创新模式。(4)进一步开展特色淡水养殖鱼类和海水养殖鱼类的人工繁育和基因编辑物质呈递技术研究。推进特色淡水养殖鱼类(包括鲈形目, 如鳜、加州鲈等)和海水养殖鱼类(大黄鱼、石斑鱼和卵形鲳参等)批量基因编辑技术研究, 改善其稳定性和高效性。从而推动基因编辑辅助育种在特色淡水养殖鱼类和海水养殖鱼类的性状改良的应用。(5)研发养殖鱼类繁育高效调控制剂。在对鱼类生殖、生理学研究基础上, 推动鱼类繁育活性物质的合成、制剂和使用剂量和方法等研究。推进制剂生产、药效和安全论证, 最终获得兽药审批, 并市场化应用。
实现鱼类苗种的“可控不育”, 不仅有利于生态风险控制, 也可以实现对鱼类苗种供应的种质管控, 从而为保障种质质量和供应提供有效的技术手段, 有效实现育种科研工作知识产权的保护, 真正推进我国水产种业的市场化、规模化发展, 推进水产种业科技研发向企业为创新主体新时代的到来。当然, 可能会有学者针对单基因编辑实现育性控制稳定性表示担忧, 而本文认为: (1)正如目前多数基因编辑改良性状本就采用单基因编辑方式, 凸显了许多性状可被“单基因”控制, 而实际上例如无肌间刺表型在自然群体中却极难遇到, 甚至多代定向选育也未能实现。以此为例, 对肌间刺主效控制基因runx2b进行定向突变。由此进行合理的推测, 多项研究报道的lhβ定向突变引发对雌性鲤科鱼类的繁育障碍[101—103], 在自然状态下发生的概率, 也应与人们发现天然的肌间刺主效控制基因runx2b突变实现“无肌间刺”鱼一样, 属自然条件下的极低概率事件。这样也表明, 我们以单基因进行的育性控制, 其恢复育性的自然突变概率将等同于自然发生“无肌间刺”的概率, 这也表明以此实现的生态安全防控, 将有效地获得相应性状的人为基因编辑位点, 在自然界中发生具有同样的低概率事件。(2)与目前多数基因编辑对象提出的“受控水体”进行养殖的生态风险防控手段, 那么, 在具有同等逃逸可能性的前提下或已经发生了逃逸事件时, “不育”的编辑个体, 发生极低概率的“可育”恢复突变, 显然较每个“可育”的基因编辑个体的逃逸, 无疑具有无法比拟的生态安全性。因此, 提示此类采用我们的单基因控制育性技术, 个别逃逸个体在自然界恰好通过自然突变发生了靶向人工修饰位点修复的概率, 应为极低概率事件, 并本就与自然界中鱼类的天然突变概率并无二致(甚至更低)。另一方面, 本技术具有的种质资源管控、保护知识产权收益、激发企业作为研发主体的创新活力等优势, 也是“可育”的基因编辑群体所无法比拟的。当然, 任何经过基因编辑的性状(包括不育)都需要研究人员和有关主管部门进行完善的、全面的生物安全程序评估后, 基于获得的实验数据, 最终做出科学的评价。
-
表 1 我国利用基因编辑技术开展的养殖鱼类生产性状改良研究报道
Table 1 Improved traits of farmed fish utilizing genome editing in China
养殖对象Farmed fish 突变基因Mutant gene 性状与改良效果Improved trait 年度Year 参考文献Reference 黄颡鱼
Pelteobagrus fulvidracomstnb F1代生长至210日龄, 体重增加137% 2011, 2014, 2020 [51—53] 尼罗罗非鱼
Oreochromis niloticusamhy, mstnb F0代突变体显示amhy是尼罗罗非鱼雄性决定的必要基因
F2代缺失mstnb纯合子雄鱼相较对照组体重增加49.45%2015, 2023 [54, 55] 半滑舌鳎
Cynoglossus semilaevisdmrt1 F0代突变体显示dmrt1是雄性决定基因 2017 [56] 团头鲂
Megalobrama amblycephalamstna, mstnb F0代杂合子分析, mstna突变体体重增加7.98%; mstnb突变体体重增加6.79% 2020 [57] 异育银鲫
Carassius gibeliopik3r1 F2代杂合突变品系分析, 年度生长提升10%以上, 饲料转化率提升10%左右 2021 [58] 泥鳅
Misgurnus anguillicaudatusmstn F0代突变体促进生长可达34.9% 2021 [59] 异育银鲫Carassius gibelio foxl2, gsdf,
cyp19a1a利用各类突变体, 研究了多倍体中各基因的不同同源基因亚型在繁育调控中的功能分化 2021, 2022 [60, 61] 黄河鲤Cyprinus carpio cyp17a1 F2代纯合子分析, 全雌单性别黄河鲤群体较同塘混养群体年度生长提升20%以上 2022 [62] 南方鲇Silurus meridionalis amhr2y F0代突变体证实amhr2y具有雄性决定功能 2022 [63] 团头鲂
Megalobrama amblycephalarunx2b F2代纯合子分析, 完全消除纯合突变的团头鲂肌间骨 2023 [64] 异育银鲫Carassius gibelio runx2b 完全敲除的纯合突变的异育银鲫肌间骨 2023 [65] 鲫Carassius auratus bmp6 F2代纯合子分析, 完全消除纯合突变的鲫肌间骨 2023 [46] 黄鳝Monopterus albus cyp19a1a, foxl2 F0代突变体显示cyp19a1a, foxl2基因对卵巢发育的调控影响 2023 [66] -
[1] FAO. The State of World Fisheries and Aquaculture 2024 – Blue Transformation in Action [M]. Rome: FAO, 2024: 11-12.
[2] Horvath P, Barrangou R. CRISPR/Cas, the immune system of bacteria and archaea [J]. Science, 2010, 327(5962): 167-170. doi: 10.1126/science.1179555
[3] Fu Y F, Foden J A, Khayter C, et al. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells [J]. Nature Biotechnology, 2013, 31(9): 822-826. doi: 10.1038/nbt.2623
[4] Laughery M F, Wyrick J J. Simple CRISPR-Cas9 genome editing in Saccharomyces cerevisiae [J]. Current Protocols in Molecular Biology, 2019, 129(1): e110. doi: 10.1002/cpmb.110
[5] Ibrahim M A, Griko, N, Junker, M, et al. Bacillus thuringiensis: a genomics and proteomics perspective [J]. Bioengineered Bugs, 2010, 1 (1): 31–50.
[6] Gratz S J, Rubinstein C D, Harrison M M, et al. CRISPR-Cas9 genome editing in Drosophila [J]. Current Protocols in Molecular Biology, 2015( 111) : 31.32. 31–31.32. 20.
[7] Hall B, Cho A, Limaye A, et al. Genome editing in mice using CRISPR/Cas9 technology [J]. Current Protocols in Cell Biology, 2018, 81(1): e57. doi: 10.1002/cpcb.57
[8] Dai Z R, Wang H L, Jin X, et al. Depletion of suppressor of cytokine signaling-1a causes hepatic steatosis and insulin resistance in zebrafish [J]. American Journal of Physiology Endocrinology and Metabolism, 2015, 308(10): E849-E859. doi: 10.1152/ajpendo.00540.2014
[9] Chinnadurai P, Stojsin D, Liu K, et al. Variability of CP4 EPSPS expression in genetically engineered soybean (Glycine max L. Merrill) [J]. Transgenic Research, 2018, 27(6): 511-524. doi: 10.1007/s11248-018-0092-z
[10] Carlson D F, Lancto C A, Zang B, et al. Production of hornless dairy cattle from genome-edited cell lines [J]. Nature Biotechnology, 2016, 34(5): 479-481. doi: 10.1038/nbt.3560
[11] Whitworth K M, Prather R S. Gene editing as applied to prevention of reproductive porcine reproductive and respiratory syndrome [J]. Molecular Reproduction and Development, 2017, 84(9): 926-933. doi: 10.1002/mrd.22811
[12] Xu K, Zhou Y R, Mu Y L, et al. CD163 and pAPN double-knockout pigs are resistant to PRRSV and TGEV and exhibit decreased susceptibility to PDCoV while maintaining normal production performance [J]. eLife, 2020( 9) : e57132.
[13] de Almeida Camargo L S, Pereira J F. Genome-editing opportunities to enhance cattle productivity in the tropics [J]. CABI Agriculture and Bioscience, 2022, 3(1): 8. doi: 10.1186/s43170-022-00075-w
[14] Cuellar C J, Amaral T F, Rodriguez-Villamil P, et al. Consequences of gene editing of PRLR on thermotolerance, growth, and male reproduction in cattle [J]. FASEB Bioadvances, 2024, 6(8): 223-234. doi: 10.1096/fba.2024-00029
[15] Flórez J M, Martins K, Solin S, et al. CRISPR/Cas9-editing of KISS1 to generate pigs with hypogonadotropic hypogonadism as a castration free trait [J]. Frontiers in Genetics, 2022(13): 1078991.
[16] Zhou W J, Wan Y J, Guo R H, et al. Generation of beta-lactoglobulin knock-out goats using CRISPR/Cas9 [J]. PLoS One, 2017, 12(10): e0186056. doi: 10.1371/journal.pone.0186056
[17] Wei J, Wagner S, Maclean P, et al. Cattle with a precise, zygote-mediated deletion safely eliminate the major milk allergen beta-lactoglobulin [J]. Scientific Reports, 2018, 8(1): 7661. doi: 10.1038/s41598-018-25654-8
[18] Tara A, Singh P, Gautam D, et al. CRISPR-mediated editing of β-lactoglobulin (BLG) gene in buffalo [J]. Scientific Reports, 2024, 14(1): 14822. doi: 10.1038/s41598-024-65359-9
[19] Ahmad M. Plant breeding advancements with “CRISPR-Cas” genome editing technologies will assist future food security [J]. Frontiers in Plant Science, 2023(14): 1133036.
[20] Liu L, Kuang Y J, Yan F, et al. Developing a novel artificial rice germplasm for dinitroaniline herbicide resistance by base editing of OsTubA2 [J]. Plant Biotechnology Journal, 2021, 19(1): 5-7. doi: 10.1111/pbi.13430
[21] Liao S Y, Qin X M, Luo L, et al. CRISPR/Cas9-induced mutagenesis of semi-rolled leaf1, 2 confers curled leaf phenotype and drought tolerance by influencing protein expression patterns and ROS scavenging in rice (Oryza sativa L.) [J]. Agronomy, 2019, 9(11): 728. doi: 10.3390/agronomy9110728
[22] Zhang A N, Liu Y, Wang F M, et al. Enhanced rice salinity tolerance via CRISPR/Cas9-targeted mutagenesis of the OsRR22 gene [J]. Molecular Breeding, 2019(39): 47.
[23] Nonaka S, Arai C, Takayama M, et al. Efficient increase of ɣ-aminobutyric acid (GABA) content in tomato fruits by targeted mutagenesis [J]. Scientific Reports, 2017, 7(1): 7057. doi: 10.1038/s41598-017-06400-y
[24] Duarte Sagawa C H, Assis R d A B, Zaini P A. Regulatory framework of CRISPR-edited crops in the United States. Pages 167-195 [M]//Abd-Elsalam K A, Ahmad A (Eds.), Global Regulatory Outlook for CRISPRized Plants. London: Academic Press, 2024.
[25] FDA. FDA Approves First-of-its-Kind Intentional Genomic Alteration in Line of Domestic Pigs for Both Human Food, Potential Therapeutic Uses [EB]. 2020.
[26] 中华人民共和国农业农村部. 2023年农业转基因生物安全证书批准清单 (2023-04-28 EB]. 2023.
[27] 中华人民共和国农业农村部. 2023年农业转基因生物安全证书批准清单(三) (2024-01-18 EB]. 2024.
[28] 中华人民共和国农业农村部. 2024年农业转基因生物安全证书批准清单(一) (2024-05-08 EB]. 2024.
[29] Blix T B, Dalmo R A, Wargelius A, et al. Genome editing on finfish: Current status and implications for sustainability [J]. Reviews in Aquaculture, 2021, 13(4): 2344-2363. doi: 10.1111/raq.12571
[30] Gao Y P, Dai Z R, Shi C, et al. Depletion of myostatin b promotes somatic growth and lipid metabolism in zebrafish [J]. Frontiers in Endocrinology, 2016(7): 88.
[31] Shi C, Lu Y, Zhai G, et al. Hyperandrogenism in POMCa-deficient zebrafish enhances somatic growth without increasing adiposity [J]. Journal of Molecular Cell Biology, 2020, 12(4): 291-304. doi: 10.1093/jmcb/mjz053
[32] Yang Z, Wong J, Wang L, et al. pomc knockout increases growth in zebrafish [J]. Aquaculture, 2023(574): 739707.
[33] Zhu J J, Liu X, Cai X L, et al. Zebrafish prmt3 negatively regulates antiviral responses [J]. Faseb Journal, 2020, 34(8): 10212-10227. doi: 10.1096/fj.201902569R
[34] Song Y A, Fan S J, Zhang D W, et al. Zebrafish maoc1 attenuates spring viremia of carp virus propagation by promoting autophagy-lysosome-dependent degradation of viral phosphoprotein [J]. Journal of Virology, 2023, 97(2): e0133822. doi: 10.1128/jvi.01338-22
[35] Liao Q, Zhu C C, Sun X Y, et al. Disruption of sirtuin 7 in zebrafish facilitates hypoxia tolerance [J]. The Journal of Biological Chemistry, 2023, 299(8): 105074. doi: 10.1016/j.jbc.2023.105074
[36] Shi L L, Zhang A X, Liu H, et al. Deletion of the foxO4 gene increases hypoxia tolerance in zebrafish [J]. International Journal of Molecular Sciences, 2023, 24(10): 8942. doi: 10.3390/ijms24108942
[37] Ou Y, Li H L, Li J, et al. Formation of different polyploids through disrupting meiotic crossover frequencies based on cntd1 knockout in zebrafish [J]. Molecular Biology and Evolution, 2024, 41(3): msae047. doi: 10.1093/molbev/msae047
[38] Slanchev K, Stebler J, de la Cueva-Méndez G, et al. Development without germ cells: the role of the germ line in zebrafish sex differentiation [J]. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(11): 4074-4079.
[39] Rodríguez-Marí A, Cañestro C, Bremiller R A, et al. Sex reversal in zebrafish fancl mutants is caused by Tp53-mediated germ cell apoptosis [J]. PLoS Genetics, 2010, 6(7): e1001034. doi: 10.1371/journal.pgen.1001034
[40] Dai X Y, Jin X, Chen X W, et al. Sufficient numbers of early germ cells are essential for female sex development in zebrafish [J]. PLoS One, 2015, 10(2): e0117824. doi: 10.1371/journal.pone.0117824
[41] Mei J, Gui J F. Genetic basis and biotechnological manipulation of sexual dimorphism and sex determination in fish [J]. Science China Life Sciences, 2015, 58(2): 124-136. doi: 10.1007/s11427-014-4797-9
[42] Zhai G, Shu T T, Xia Y G, et al. Characterization of sexual trait development in cyp17a1-deficient zebrafish [J]. Endocrinology, 2018, 159(10): 3549-3562. doi: 10.1210/en.2018-00551
[43] Yan Y L, Batzel P, Titus T, et al. A hormone that lost its receptor: anti-müllerian hormone (AMH) in zebrafish gonad development and sex determination [J]. Genetics, 2019, 213(2): 529-553. doi: 10.1534/genetics.119.302365
[44] Romano S, Kaufman O H, Marlow F L. Loss of dmrt1 restores zebrafish female fates in the absence of cyp19a1a but not rbpms2a/b [J]. Development, 2020, 147(18): dev190942.
[45] Ruan Y L, Li X H, Wang X Y, et al. New insights into the all-testis differentiation in zebrafish with compromised endogenous androgen and estrogen synthesis [J]. PLoS Genetics, 2024, 20(3): e1011170. doi: 10.1371/journal.pgen.1011170
[46] Kuang Y Y, Zheng X H, Cao D C, et al. Generate a new crucian carp (Carassius auratus) strain without intermuscular bones by knocking out bmp6 [J]. Aquaculture, 2023, 569: 739407. doi: 10.1016/j.aquaculture.2023.739407
[47] Nie C H, Wan S M, Chen Y L, et al. Single-cell transcriptomes and runx2b–/- mutants reveal the genetic signatures of intermuscular bone formation in zebrafish [J]. National Science Review, 2022, 9(11): nwac152. doi: 10.1093/nsr/nwac152
[48] Edvardsen R B, Leininger S, Kleppe L, et al. Targeted mutagenesis in Atlantic salmon (Salmo salar L.) using the CRISPR/Cas9system induces complete knockout individuals in the F0 generation [J]. PLoS One, 2014, 9(9): e108622. doi: 10.1371/journal.pone.0108622
[49] Li M H, Yang H H, Zhao J U, et al. Efficient and heritable gene targeting in tilapia by CRISPR/Cas9 [J]. Genetics, 2014, 197(2): 591-599. doi: 10.1534/genetics.114.163667
[50] Moran M N, Jones D B, Jensen S A, et al. Optimising commercial traits through gene editing in aquaculture: Strategies for accelerating genetic improvement [J]. Reviews in Aquaculture, 2024, 16(3): 1127-1159. doi: 10.1111/raq.12889
[51] Dong Z J, Ge J C, Li K, et al. Heritable targeted inactivation of myostatin gene in yellow catfish (Pelteobagrus fulvidraco) using engineered zinc finger nucleases [J]. PLoS One, 2011, 6(12): e28897. doi: 10.1371/journal.pone.0028897
[52] Dong Z J, Ge J C, Xu Z Q, et al. Generation of myostatin B knockout yellow catfish (Tachysurus fulvidraco) using transcription activator-like effector nucleases [J]. Zebrafish, 2014, 11(3): 265-274. doi: 10.1089/zeb.2014.0974
[53] Zhang X C, Wang F, Dong Z J, et al. A new strain of yellow catfish carrying genome edited myostatin alleles exhibits double muscling phenotype with hyperplasia [J]. Aquaculture, 2020(523): 735187.
[54] Li M H, Sun Y L, Zhao J E, et al. A tandem duplicate of anti-müllerian hormone with a missense SNP on the Y chromosome is essential for male sex determination in nile tilapia, Oreochromis niloticus [J]. Plos Genetics, 2015, 11(11): e1005678. doi: 10.1371/journal.pgen.1005678
[55] Wu Y, Wu T F, Yang L Y, et al. Generation of fast growth nile tilapia (Oreochromis niloticus) by myostatin gene mutation [J]. Aquaculture, 2023(562): 738762.
[56] Cui Z K, Liu Y, Wang W W, et al. Genome editing reveals dmrt1 as an essential male sex-determining gene in Chinese tongue sole (Cynoglossus semilaevis) [J]. Scientific Reports, 2017(7): 42213.
[57] Sun Y, Zheng G D, Nissa M, et al. Disruption of mstna and mstnb gene through CRISPR/Cas9 leads to elevated muscle mass in blunt snout bream (Megalobrama amblycephala) [J]. Aquaculture, 2020(528): 735597.
[58] Huang J F, Shi C, Gao Y P, et al. Heterozygous depletion of pik3r1 improves growth and feed conversion efficiency in gibel carp (Carassius gibelio) [J]. Aquaculture, 2021(545): 737207.
[59] Tao B B, Tan J, Chen L, et al. CRISPR/Cas9system-based myostatin-targeted disruption promotes somatic growth and adipogenesis in loach, Misgurnus anguillicaudatus [J]. Aquaculture, 2021(544): 737097.
[60] Gan R H, Wang Y, Li Z, et al. Functional divergence of multiple duplicated foxl2 homeologs and alleles in a recurrent polyploid fish [J]. Molecular Biology and Evolution, 2021, 38(5): 1995-2013. doi: 10.1093/molbev/msab002
[61] Wang M T, Li Z, Ding M, et al. Two duplicated gsdf homeologs cooperatively regulate male differentiation by inhibiting cyp19a1a transcription in a hexaploid fish [J]. PLoS Genetics, 2022, 18(6): e1010288. doi: 10.1371/journal.pgen.1010288
[62] Zhai G, Shu T T, Chen K X, et al. Successful production of an all-female common carp (Cyprinus carpio L. ) population using cyp17a1-deficient neomale carp [J]. Engineering, 2022(8): 181-189.
[63] Zheng S Q, Tao W J, Yang H W, et al. Identification of sex chromosome and sex-determining gene of southern catfish (Silurus meridionalis) based on XX, XY and YY genome sequencing [J]. Proceedings Biological Sciences, 2022, 289(1971): 20212645.
[64] Dong Q, Nie C H, Wu Y M, et al. Generation of blunt snout bream without intermuscular bones by runx2b gene mutation [J]. Aquaculture, 2023(567): 739263.
[65] Gan R H, Li Z, Wang Z W, et al. Creation of intermuscular bone-free mutants in amphitriploid gibel carp by editing two duplicated runx2b homeologs [J]. Aquaculture, 2023(567): 739300.
[66] Hou M X, Feng K, Luo H R, et al. Multi-locus gene editing effectively knocked out cyp19a1a and foxl2 in Monopterus albus, a hermaphroditic fish [J]. Aquaculture, 2023(565): 739130.
[67] Giraldo P A, Shinozuka H, Spangenberg G C, et al. Safety assessment of genetically modified feed: is there any difference from food [J]? Frontiers in Plant Science, 2019( 10) : 1592.
[68] Goodman R E, Panda R, Ariyarathna H. Evaluation of endogenous allergens for the safety evaluation of genetically engineered food crops: review of potential risks, test methods, examples and relevance [J]. Journal of Agricultural and Food Chemistry, 2013, 61(35): 8317-8332. doi: 10.1021/jf400952y
[69] Krutz N L, Kimber I, Winget J, et al. Application of AllerCatPro 2.0 for protein safety assessments of consumer products [J]. Frontiers in allergy, 2023(4): 1209495.
[70] 张志伟, 曹哲明, 杨弘, 等. 草鱼野生和养殖群体间遗传变异的微卫星分析 [J]. 动物学研究, 2006, 27(2): 189-196.] doi: 10.3321/j.issn:0254-5853.2006.02.010 Zhang Z W, Cao Z M, Yang H, et al. Microsatellites analysis on genetic variation between wild and cultured populations of Ctenopharyngodon idella [J]. Zoological Research, 2006, 27(2): 189-196. [ doi: 10.3321/j.issn:0254-5853.2006.02.010
[71] Gutierrez A P, Yáñez J M, Davidson W S. Evidence of recent signatures of selection during domestication in an Atlantic salmon population [J]. Marine genomics, 2016(26): 41-50.
[72] Gutierrez A P, Yáñez J M, Fukui S, et al. Genome-wide association study (GWAS) for growth rate and age at sexual maturation in Atlantic salmon (Salmo salar) [J]. PLoS One, 2015, 10(3): e0119730. doi: 10.1371/journal.pone.0119730
[73] Karlsson S, Diserud O H, Fiske P, et al. Widespread genetic introgression of escaped farmed Atlantic salmon in wild salmon populations [J]. ICES Journal of Marine Science, 2016, 73(10): 2488-2498. doi: 10.1093/icesjms/fsw121
[74] Jensen Ø, Dempster T, Thorstad E, et al. Escapes of fish from Norwegian sea-cage aquaculture: causes, consequences, and prevention [J]. Aquaculture Environment Interactions, 2010(1): 71-83.
[75] Valosaari K R, Aikio S, Kaitala V. Male mating strategy and the introgression of a growth hormone transgene [J]. Evolutionary Applications, 2008, 1(4): 608-619. doi: 10.1111/j.1752-4571.2008.00046.x
[76] Kinlan B P, Gaines S D. Propagule dispersal in marine and terrestrial environments: A community perspective [J]. Ecology, 2003, 84(8): 2007-2020. doi: 10.1890/01-0622
[77] Dempster T, Arechavala-Lopez P, Barrett L T, et al. Recapturing escaped fish from marine aquaculture is largely unsuccessful: alternatives to reduce the number of escapees in the wild [J]. Reviews in Aquaculture, 2018, 10(1): 153-167. doi: 10.1111/raq.12153
[78] Ju R T, Li X, Jiang J J, et al. Emerging risks of non-native species escapes from aquaculture: Call for policy improvements in China and other developing countries [J]. Journal of Applied Ecology, 2020, 57(1): 85-90. doi: 10.1111/1365-2664.13521
[79] Yang M, Tian C, Liang X F, et al. Genetic structure and diversity in natural and stocked populations of the mandarin fish (Siniperca chuatsi) in China [J]. Genetics and Molecular Research, 2015, 14(2): 5153-5160. doi: 10.4238/2015.May.18.5
[80] Toledo-Guedes K, Atalah J, Izquierdo-Gomez D, et al. Domesticating the wild through escapees of two iconic mediterranean farmed fish species [J]. Scientific Reports, 2024, 14(1): 23772. doi: 10.1038/s41598-024-74172-3
[81] Berstad A J, Fludal Heimstad L. Experience from Introduction of the Design Code NS 9415 to the Aquaculture Industry in Norway and Expanding the Scope to Cover Also Operations [C]. ASME 2017 36th International Conference on Ocean, Offshore and Arctic Engineering. Trondheim, Norway, 2017: V009T012A003.
[82] Glover K A, Solberg M F, McGinnity P, et al. Half a century of genetic interaction between farmed and wild Atlantic salmon: Status of knowledge and unanswered questions [J]. Fish and Fisheries, 2017, 18(5): 890-927. doi: 10.1111/faf.12214
[83] Waltz E. GABA-enriched tomato is first CRISPR-edited food to enter market [J]. Nature Biotechnology, 2022, 40(1): 9-11. doi: 10.1038/d41587-021-00026-2
[84] Gratacap R L, Wargelius A, Edvardsen R B, et al. Potential of genome editing to improve aquaculture breeding and production [J]. Trends in Genetics, 2019, 35(9): 672-684. doi: 10.1016/j.tig.2019.06.006
[85] Hallerman E M, Bredlau J P, Camargo L S A, et al. Towards progressive regulatory approaches for agricultural applications of animal biotechnology [J]. Transgenic Research, 2022, 31(2): 167-199. doi: 10.1007/s11248-021-00294-3
[86] Hartnell G F, Cromwell G L, Dana G R, et al. Best Practices for the Conduct of Animal Studies to Evaluate Crops Genetically Modified for Output Traits [M]. Washington, DC: International Life Sciences Institute, 2007.
[87] Chen G H, Huang J F, Jia J Y, et al. The food safety assessment of all-female common carp (Cyprinus carpio) (cyp17a1+/-; XX genotype) generated using genome editing technology [J]. Food and Chemical Toxicology, 2023(181): 114103.
[88] Robinson N A, Østbye T-K K, Kettunen A H, et al. A guide to assess the use of gene editing in aquaculture [J]. Reviews in Aquaculture, 2024, 16(2): 775-784. doi: 10.1111/raq.12866
[89] 徐旭丹, 刘强, 黄伟, 等. 外来养殖鱼类的入侵风险评估及防控对策——以尼罗罗非鱼为例 [J]. 生物安全学报, 2022, 31(3): 278-288.] Xu X D, Liu Q, Huang W, et al. Risk assessment and control of the exotic farmed fish, the Nile tilapia (Oreochromis niloticus) [J]. Journal of Biosafety, 2022, 31(3): 278-288. [
[90] Wong T T, Zohar Y. Production of reproductively sterile fish: A mini-review of germ cell elimination technologies [J]. General and Comparative Endocrinology, 2015(221): 3-8.
[91] Hu W, Zhu Z Y. Integration mechanisms of transgenes and population fitness of GH transgenic fish [J]. Science China Life Sciences, 2010, 53(4): 401-408. doi: 10.1007/s11427-010-0088-2
[92] Wargelius A. Application of genome editing in aquatic farm animals: Atlantic salmon [J]. Transgenic Research, 2019, 28(Suppl 2): 101-105.
[93] Xu L, Zhao M L, Ryu J H, et al. Reproductive sterility in aquaculture: A review of induction methods and an emerging approach with application to Pacific Northwest finfish species [J]. Reviews in Aquaculture, 2023, 15(1): 220-241. doi: 10.1111/raq.12712
[94] Roy S, Kumar V, Behera B, et al. CRISPR/Cas genome editing—can it become a game changer in future fisheries sector [J]? Frontiers in Marine Science, 2022(9): 924475.
[95] Yu F, Xiao J, Liang X Y, et al. Rapid growth and sterility of growth hormone gene transgenic triploid carp [J]. Chinese Science Bulletin, 2011, 56(16): 1679-1684. doi: 10.1007/s11434-011-4446-7
[96] Zhang C, Li Q, Zhu L, et al. Abnormal meiosis in fertile and sterile triploid cyprinid fish [J]. Science China Life Sciences, 2021, 64(11): 1917-1928. doi: 10.1007/s11427-020-1900-7
[97] Lu M, Zhang Q C, Zhu Z Y, et al. An efficient approach to synthesize sterile allopolyploids through the combined reproduction mode of ameiotic oogenesis and sperm-egg fusion in the polyploid Carassius complex [J]. Science Bulletin, 2023, 68(10): 1038-1050. doi: 10.1016/j.scib.2023.04.029
[98] 鲁蒙, 周莉, 桂建芳. “无减数融合”生殖方式的发现及其育种实践意义 [J]. 科学通报, 2024, 69(19): 2704-2706.] Lu M, Zhou L, Gui J F, et al. Discovery of ameio-fusiongenesis and its breeding applications [J]. Chinese Science Bulletin, 2024, 69(19): 2704-2706. [
[99] Zhang Y S, Chen J, Cui X J, et al. A controllable on-off strategy for the reproductive containment of fish [J]. Scientific Reports, 2015(5): 7614.
[100] Güralp H, Skaftnesmo K O, Kjærner-Semb E, et al. Rescue of germ cells in dnd crispant embryos opens the possibility to produce inherited sterility in Atlantic salmon [J]. Scientific Reports, 2020, 10(1): 18042. doi: 10.1038/s41598-020-74876-2
[101] Chu L H, Li J Z, Liu Y, et al. Targeted gene disruption in zebrafish reveals noncanonical functions of LH signaling in reproduction [J]. Molecular Endocrinology, 2014, 28(11): 1785-1795. doi: 10.1210/me.2014-1061
[102] Shang G H, Peng X Y, Ji C, et al. Steroidogenic acute regulatory protein and luteinizing hormone are required for normal ovarian steroidogenesis and oocyte maturation in zebrafish [J]. Biology of Reproduction, 2019, 101(4): 760-770. doi: 10.1093/biolre/ioz132
[103] Shi S C, Zhang Y Q, Huang J F, et al. Effective “off-on” switch for fertility control in female zebrafish [J]. Frontiers in Marine Science, 2024(11): 1381305.



 下载:
下载: